清华大学脑与智能实验室及生物医学工程系研究员、智源研究员宋森等针对预测蛋白质互相作用问题,提出了采用图神经网络,先在单个蛋白上通过从对微扰动的蛋白结构预测其原始蛋白结构这一自监督学习任务,学习蛋白质结构的几何知识的方法,基于该网络学到的表征训练的模型在预测氨基酸突变对蛋白质相互结合强度的影响这一任务中明显好于其他模型,尤其在多个氨基酸突变的情况下表现尤其突出;相关成果发表在《PLOS 计算生物学》杂志上(2021年6月)
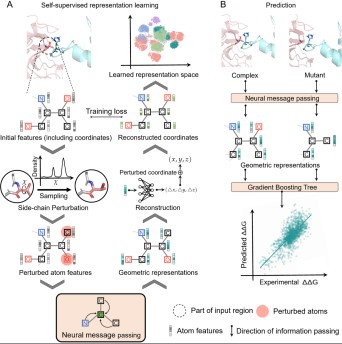
图. 通过自监督方法学习蛋白几何结构(左)可以帮助预测突变对蛋白间结合强度的预测(右)
(图片来源:学者提供)
Xianggen Liu, Yunan Luo, Pengyong Li, Sen Song*, Jian Peng*,Deep geometric representations for modeling effects of mutations on protein-protein binding affinity. PLoS Computational Biology 17.8 (2021): e1009284
